前言
肿瘤非整倍体事件,即整个染色体或染色体臂的拷贝数改变,是癌症的一个标志,其驱动力主要取决于细胞和基因组环境。 全基因组加倍(whole genome duplication, WGD)是一种宏观进化的遗传改变,影响约三分之一的人类肿瘤。 WGD在原核生物和真核生物中都有研究,从进化的角度来看,经历过WGD的生物具有一个优势,使它们能够战胜二倍体的祖先。在正常的人类发育中,基因组的四倍化是罕见的,除了在减数分裂期间的生殖细胞。WGD通过进一步传播基因组不稳定性来促进肿瘤发生,从而为肿瘤进化创造多样化的底物,减弱对必需基因突变的选择,并增加对染色体错误分离的耐受性。 WGD与几种分子和临床肿瘤特征有关:包括TP53突变,更高的突变负荷,增加的增殖特征和更差的总生存期。在促进肿瘤发生的同时,WGD还与癌细胞中细胞脆弱性的改变有关,例如对有丝分裂驱动蛋白KIF18A抑制的敏感性增加 [1,2] 。 在2004年,就有研究者从多倍体、非整倍体、基因组的不稳定性和癌症的相关性方面进行回顾分析,并提出倍性特异性的致死性可以被用于识别开发抗肿瘤药物的新靶点 [3] 。
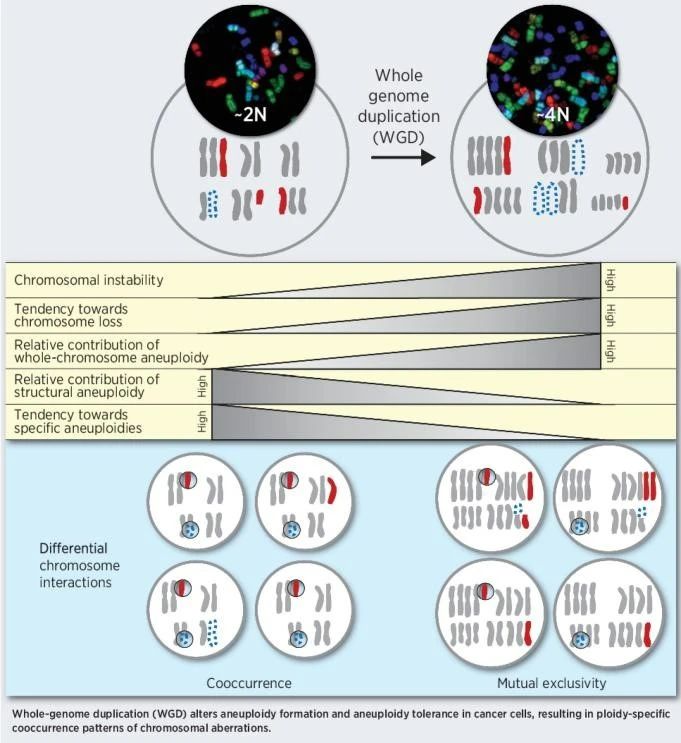
首先,增殖的WGD+细胞在基因组上不稳定,并迅速积累数量和结构上的染色体异常;
这些特征使新生的WGD+肿瘤细胞在存在其他致命的基因组改变的情况下增殖,同时对增加的基因排列进行取样,最终使表型跳跃导致肿瘤。 WGD的发病率因癌症类型而显著不同,影响58%的生殖细胞肿瘤,而只有5%或更少的非霍奇金淋巴瘤和胃肠道神经内分泌肿瘤 [1] 。在不同肿瘤类型中,WGD-和WGD+肿瘤中存在不同的非整倍体模式,WGD+肿瘤的非整倍体明显高于WGD-肿瘤,且WGD+肿瘤在整个基因组中的非整倍体分布更均匀 [2] 。
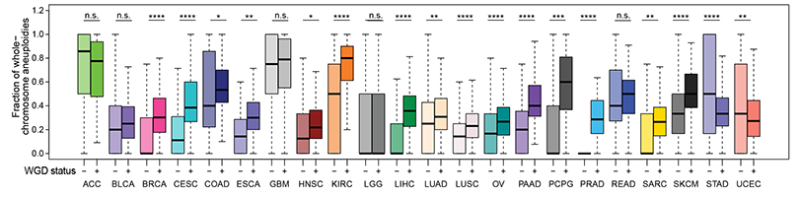
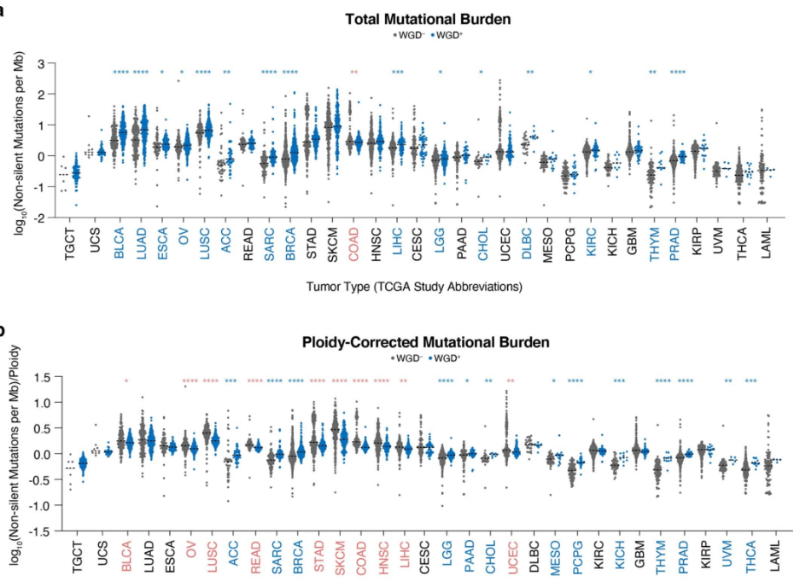
WGD+肿瘤的遗传分析显示约36%的肿瘤在其进化过程中经历了至少一次WGD,且不同的肿瘤亚型中WGD的发生存在较大差异,因此特定的遗传、生理或微环境可以促进或抑制WGD驱动的肿瘤发生 [4] 。WGD+肿瘤往往具有更 高的总突变负荷。然而,一些肿瘤亚型中,经倍性校正的WGD-肿瘤比WGD+肿瘤具有更高的突变负荷,这种情况往往发生在具有高突变负荷的亚型中,其特征是易于发生微卫星不稳定性(MSI)或暴露于外源性诱变剂。在突变负荷较低的亚型组中,具有更高的倍性校正突变负荷的却是WGD+肿瘤。
具有MSI或POLE基因突变的肿瘤,具有非常高的突变负担,往往不会发生WGD事件。
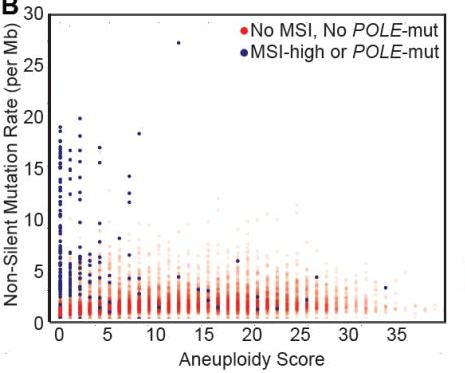
图4. 非整倍体评分与MSI+POLE分析 [5]
在乳腺癌中,WGD+患者具有相对较高的HRD评分的特征,在每个亚型中,WGD+肿瘤的HRD评分高于WGD-肿瘤,但WGD+肿瘤在不同亚型之间没有观察到差异。 WGD和HRD与特定的indel模式共存,其中对替代同源重组修复过程的进一步基因组特征显示,indel信号比SNV更好、更可靠地预测WGD连锁HRD表型。在三阴性乳腺癌队列中,35.7%的患者经常伴有HRD,而28.6%的患者同时伴有WGD+。同样,在HR+/HER2-和HER2+队列中,有WGD的HRD病例比无WGD的HRD病例更多,而在HR+/HER2+肿瘤中,没有观察到差异 [6] 。
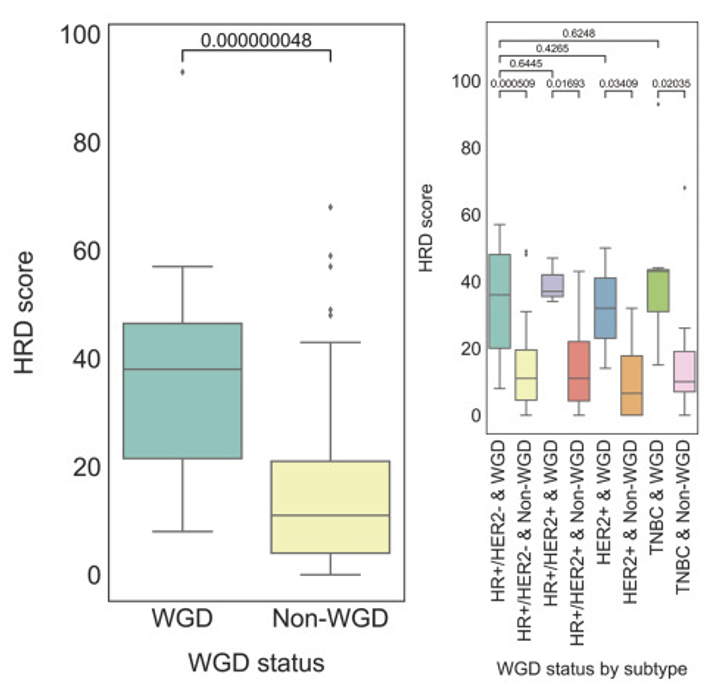
图5. HRD与WGS相关性 [6]

TP53肿瘤抑制因子在WGD中的作用一直是个谜:p53可以阻断四倍体细胞的增殖,作为WGD的屏障,但也可以促进有丝分裂旁路,这是WGD通过核内复制的关键步骤 。在野生型(WT)p53肿瘤中,WGD经常与E2F通路的激活相关,特别是编码细胞周期蛋白E1(Cyclin E1)的CCNE1的扩增。研究者发现细胞周期蛋白E1表达的升高会导致复制性应激,从而激活ATR-和chk1依赖的G2期阻滞。p53通过其下游靶点p21与Wee1一起有效抑制有丝分裂周期蛋白依赖性激酶的活性,激活APC/CCdh1,促进有丝分裂旁路。细胞周期蛋白E的表达抑制有丝分裂旁路后p53依赖的衰老,允许细胞完成核内复制,因此研究结果表明,p53可以通过促进WGD来促进癌症的进化 [7]。
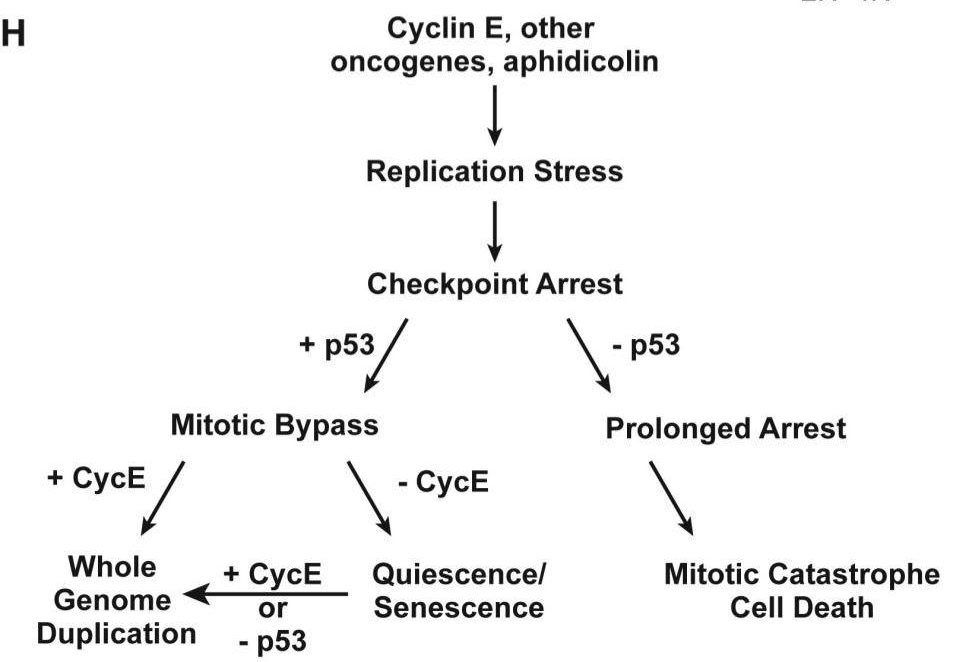
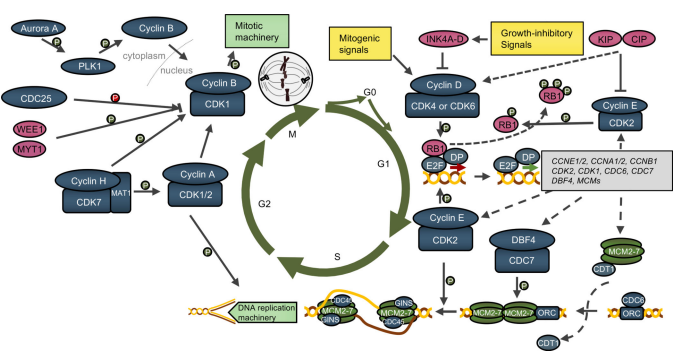
图8. 细胞周期 [9]
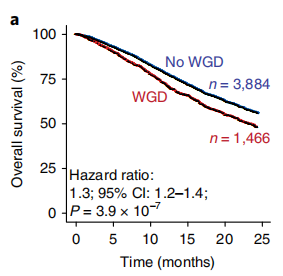
在一项万人前瞻性临床测序的转移性研究中,研究者发现该队列的特征为WGD+在晚期疾病环境中的提示总体生存率更差,在调整了癌症类型、年龄和TP53突变状态后,WGD仍然与泛癌患者总生存率的下降显著相关。
在另一项研究中,研究者利用全基因组泛癌症分析(PCAWG)和癌症基因组图谱的测序数据来阐明WGD影响预后的机制。 研究发现WGD与几个因素相关,如LOH区域的长度。使用WGD相关因素进行的生存分析显示,在WGD+的样本(WGD)和WGD-的样本(nWGD)中,chr17中较长的LOH区域和LOH与不良预后相关。除这两个因素外,nWGD样本显示肿瘤抑制基因(TSGs)的突变数量与预后相关。据此,研究者进一步提出了WGD进化模型。
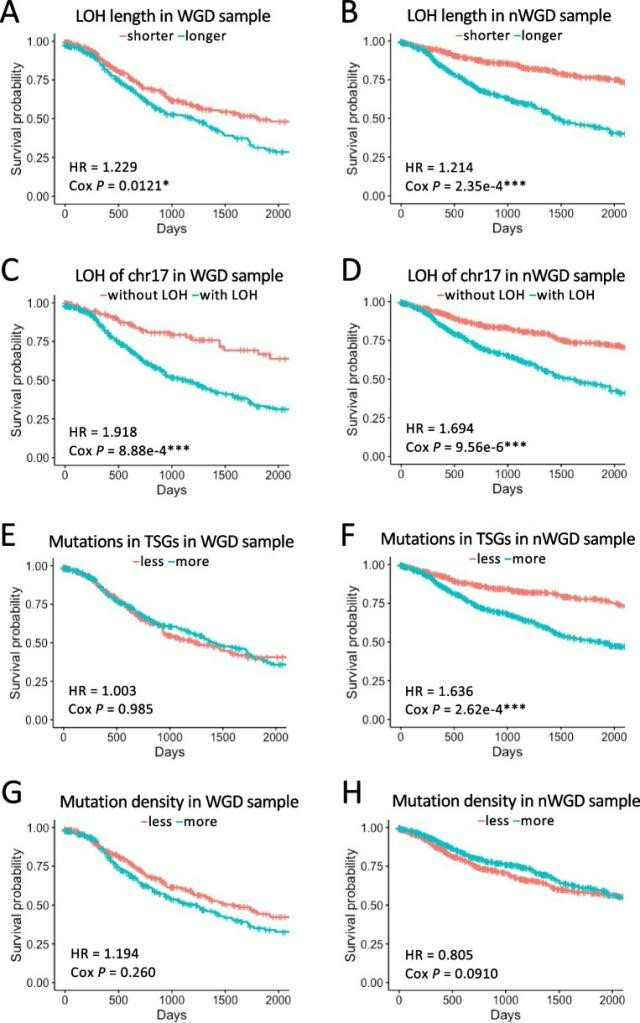
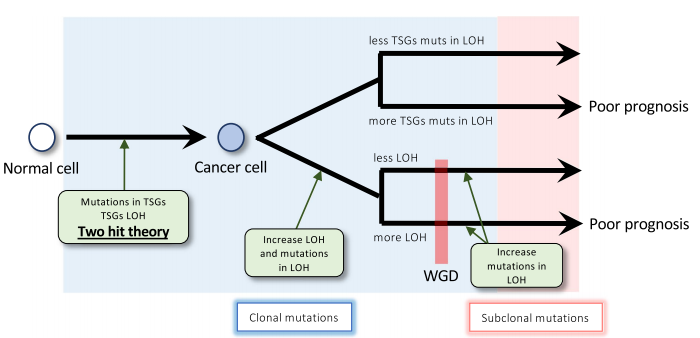
图10-11. WGD相关因素的生存分析及WGD进化模型 [10]
WGD作为一个潜在的生物标志物,目前可以选择WGS、WES、SNParray等多种方法。对WGD阳性的判定主要采用 肿瘤倍性(ploidy)值 和 染色体不稳定性(Chromosomal instability,CIN)值 来判断。
采用肿瘤倍性判断WGD,是基于一项对CCLE细胞系非整倍性研究 。研究者基于前期CCLE细胞系的WGD状态和染色体臂非整倍体景观,确定细胞系倍性为<2.5,分类为WGD−,倍性为>3,分类为WGD+ [2,11] 。同时引入了非整倍体评分(Aneuploidy score, AS)的概念。
非整倍体评分被定义为获得或丢失的染色体臂的总数,反映了一个样本中具有臂水平拷贝数改变的染色体臂的总数。 在最新的研究中,这种非整倍体评分范围从0到39,即人类常染色体染色体臂的总数:1-12和16-20染色体的p和q臂,以及端中心染色体13-15和21-22染色体的q臂 [5] 。
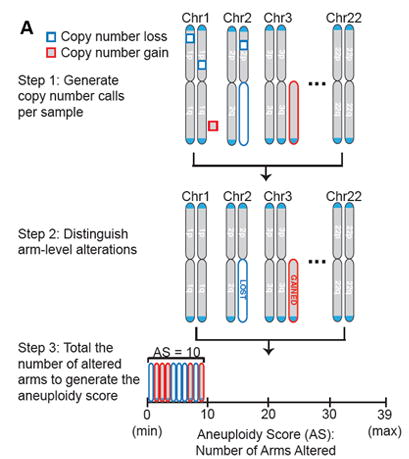
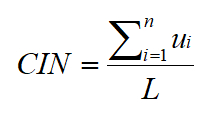
图13. 公式来源 [6]
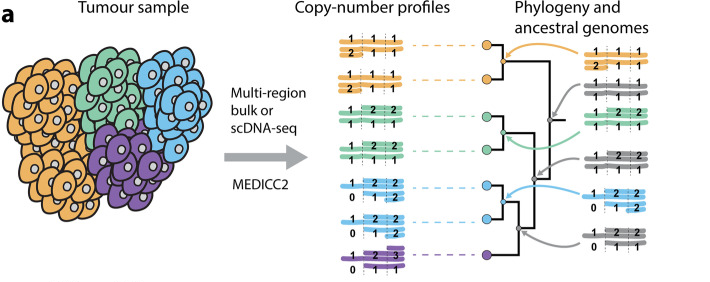
图14. MEDICC2算法 [12]
WGD+肿瘤细胞的特异性遗传敏感性做为治疗新靶标的研究工作正在逐渐展开,2023年美国癌症研究协会(AACR)年会,来自多个团队的研究已经确定有丝分裂驱动蛋白KIF18A对CIN和非整倍体细胞的增殖具有选择性必需。 通过基因靶向KIF18A和新型小分子抑制剂,多项研究数据支持KIF18A作为CIN肿瘤治疗靶点 [13-16] 。
[1] Bielski CM, Zehir A, Penson AV, Donoghue MTA, Chatila W, Armenia J, Chang MT, Schram AM, Jonsson P, Bandlamudi C, Razavi P, Iyer G, Robson ME, Stadler ZK, Schultz N, Baselga J, Solit DB, Hyman DM, Berger MF, Taylor BS. Genome doubling shapes the evolution and prognosis of advanced cancers. Nat Genet. 2018 Aug;50(8):1189-1195. doi: 10.1038/s41588-018-0165-1. Epub 2018 Jul 16. PMID: 30013179; PMCID: PMC6072608.
[2] Prasad K, Bloomfield M, Levi H, Keuper K, Bernhard SV, Baudoin NC, Leor G, Eliezer Y, Giam M, Wong CK, Rancati G, Storchová Z, Cimini D, Ben-David U. Whole-Genome Duplication Shapes the Aneuploidy Landscape of Human Cancers. Cancer Res. 2022 May 3;82(9):1736-1752. doi: 10.1158/0008-5472.CAN-21-2065. PMID: 35502547; PMCID: PMC9069772.
[3] Storchova Z, Pellman D. From polyploidy to aneuploidy, genome instability and cancer. Nat Rev Mol Cell Biol. 2004 Jan;5(1):45-54. doi: 10.1038/nrm1276. PMID: 14708009.
[4] Quinton RJ, DiDomizio A, Vittoria MA, Kotýnková K, Ticas CJ, Patel S, Koga Y, Vakhshoorzadeh J, Hermance N, Kuroda TS, Parulekar N, Taylor AM, Manning AL, Campbell JD, Ganem NJ. Whole-genome doubling confers unique genetic vulnerabilities on tumour cells. Nature. 2021 Feb;590(7846):492-497. doi: 10.1038/s41586-020-03133-3. Epub 2021 Jan 27. Erratum in: Nature. 2021 May;593(7860):E15. PMID: 33505027; PMCID: PMC7889737.
[5] Taylor AM, Shih J, Ha G, Gao GF, Zhang X, Berger AC, Schumacher SE, Wang C, Hu H, Liu J, Lazar AJ; Cancer Genome Atlas Research Network; Cherniack AD, Beroukhim R, Meyerson M. Genomic and Functional Approaches to Understanding Cancer Aneuploidy. Cancer Cell. 2018 Apr 9;33(4):676-689.e3. doi: 10.1016/j.ccell.2018.03.007. Epub 2018 Apr 2. PMID: 29622463; PMCID: PMC6028190.
[6] Wu CH, Hsieh CS, Chang YC, Huang CC, Yeh HT, Hou MF, Chung YC, Tu SH, Chang KJ, Chattopadhyay A, Lai LC, Lu TP, Li YH, Tsai MH, Chuang EY. Differential whole-genome doubling and homologous recombination deficiencies across breast cancer subtypes from the Taiwanese population. Commun Biol. 2021 Sep 9;4(1):1052. doi: 10.1038/s42003-021-02597-x. PMID: 34504292; PMCID: PMC8429690.
[7] Zeng J, Hills SA, Ozono E, Diffley JFX. Cyclin E-induced replicative stress drives p53-dependent whole-genome duplication. Cell. 2023 Feb 2;186(3):528-542.e14. doi: 10.1016/j.cell.2022.12.036. Epub 2023 Jan 20. PMID: 36681079.
[8] 游路宽,佘亚军,陈初鹏.细胞周期蛋白E1与恶性肿瘤关系的研究进展[J].癌变.畸变.突变, 2016.
[9] Suski JM, Braun M, Strmiska V, Sicinski P. Targeting cell-cycle machinery in cancer. Cancer Cell. 2021 Jun 14;39(6):759-778. doi: 10.1016/j.ccell.2021.03.010. Epub 2021 Apr 22. PMID: 33891890; PMCID: PMC8206013.
[10] Kikutake C, Suyama M. Pan-cancer analysis of whole-genome doubling and its association with patient prognosis. BMC Cancer. 2023 Jul 3;23(1):619. doi: 10.1186/s12885-023-11132-6. PMID: 37400777; PMCID: PMC10318685.
[11] Cohen-Sharir Y, McFarland JM, Abdusamad M, Marquis C, Bernhard SV, Kazachkova M, Tang H, Ippolito MR, Laue K, Zerbib J, Malaby HLH, Jones A, Stautmeister LM, Bockaj I, Wardenaar R, Lyons N, Nagaraja A, Bass AJ, Spierings DCJ, Foijer F, Beroukhim R, Santaguida S, Golub TR, Stumpff J, Storchová Z, Ben-David U. Aneuploidy renders cancer cells vulnerable to mitotic checkpoint inhibition. Nature. 2021 Feb;590(7846):486-491. doi: 10.1038/s41586-020-03114-6. Epub 2021 Jan 27. PMID: 33505028; PMCID: PMC8262644.
[12] Kaufmann TL, Petkovic M, Watkins TBK, Colliver EC, Laskina S, Thapa N, Minussi DC, Navin N, Swanton C, Van Loo P, Haase K, Tarabichi M, Schwarz RF. MEDICC2: whole-genome doubling aware copy-number phylogenies for cancer evolution. Genome Biol. 2022 Nov 14;23(1):241. doi: 10.1186/s13059-022-02794-9. PMID: 36376909; PMCID: PMC9661799.
[13] Jie Jack Li, Guiping Zhang, Jiapeng Li; Abstract 4482: Discovery of GH2616, a potent and selective KIF18A inhibitor with robust in vivo efficacy in p53 mutant cancer. Cancer Res 1 April 2023; 83 (7_Supplement): 4482. https://doi.org/10.1158/1538-7445.AM2023-4482
[14] James D. Joseph, Katherine Schutt, Kira Fisher, Katelyn Queen, Olivia Budington, Weifeng Mao, Wei Liu, Zhengqing Zhu, Xiangping Zhang, Yisong Xiao, Kunmin Lai, Xiaohui Gu, Jason Stumpff, Fred Aswad; Abstract 4965: Targeting the mitotic kinesin, KIF18A, in chromosomally unstable cancers. Cancer Res 1 April 2023; 83 (7_Supplement): 4965. https://doi.org/10.1158/1538-7445.AM2023-4965
[15] Aaron F. Phillips, Rumin Zhang, Akanksha Verma, Tamar Feinberg, Mia Jaffe, Marysol Chu Carty, Ryan Schulz, Sarah Bettigole, Celia Andreu, Scott Drutman, Michael Su, Derek Cogan, Christina H. Eng; Abstract 517: Targeting chromosomally unstable tumors with a selective KIF18A inhibitor. Cancer Res 1 April 2023; 83 (7_Supplement): 517. https://doi.org/10.1158/1538-7445.AM2023-517
[16] Brian Belmontes, Jodi Moriguchi, Grace Chung, Jan Sun, Maria Stefania S. Ninniri, Kelly Hanestad, Kui Chen, John D. McCarter, Upendra P. Dahal, Sudipa Ghimire-Rijal, Yue Hao, Christopher P. Mohr, Xinchao Yu, Matthew G. Rees, Melissa Ronan, Jennifer Roth, Sheroy Minocherhomji, Jennifer R. Allen, Matthew P. Bourbeau, Paul E. Hughes, Nuria A Tamayo, Marc N. Payton; Abstract 516: Discovery and preclinical characterization of AMG650, a first-in-class inhibitor of kinesin KIF18A motor protein with potent activity against chromosomally unstable cancers. Cancer Res 1 April 2023; 83 (7_Supplement): 516. https://doi.org/10.1158/1538-7445.AM2023-516